La menace des bactéries résistantes aux antibiotiques
Selon l’OMS, alors que le monde est confronté chaque année à près de 5 millions de décès liés à des bactéries résistantes aux antibiotiques, il est extrêmement urgent de trouver des médicaments potentiels pour faire face à cette situation.
Aujourd'hui, une équipe dirigée par le pionnier de la biotechnologie César de la Fuente utilise des méthodes de calcul basées sur l'intelligence artificielle (IA) pour extraire des traits génétiques de parents humains éteints comme les Néandertaliens, afin de ramener leurs antibiotiques 40 000 ans en arrière.

Modèle de l'espèce humaine éteinte Néandertal. Photo : Getty
Grâce à des recherches, les scientifiques ont découvert un certain nombre de petites molécules protéiques ou peptidiques qui ont la capacité de combattre les bactéries, ce qui pourrait ouvrir la voie à de nouveaux médicaments pour lutter contre les infections chez l’homme.
Les antibiotiques (comme la pénicilline) sont ceux qui sont produits naturellement (par un autre micro-organisme antimicrobien), tandis que les antimicrobiens non antibiotiques (comme les sulfamides et les antiseptiques) sont ceux qui sont entièrement synthétiques.
Cependant, les deux types d'antimicrobiens ont le même objectif : tuer ou empêcher la croissance des micro-organismes, et relèvent tous deux de la chimiothérapie antimicrobienne. Les antibactériens comprennent les antiseptiques, les savons antibactériens et les détergents chimiques ; les antibiotiques, quant à eux, sont des antibactériens plus spécialisés, utilisés en médecine et parfois dans l'alimentation animale.
Les antibiotiques ne sont pas efficaces contre les virus qui causent des maladies telles que le rhume ou la grippe. Les médicaments qui inhibent les virus sont donc appelés antiviraux ou antirétroviraux, et non antibiotiques.
« Cela nous permet de découvrir de nouvelles séquences, de nouveaux types de molécules jamais observés chez les organismes vivants, ce qui nous ouvre une réflexion plus large sur la diversité moléculaire », a déclaré le Dr Cesar de la Fuente de l'Université de Pennsylvanie (États-Unis), qui a dirigé l'équipe de recherche. « Les bactéries actuelles n'ont jamais rencontré ces nouvelles molécules ; cela pourrait donc être une excellente occasion de lutter contre les pathogènes difficiles à traiter d'aujourd'hui. »
Les experts affirment que de nouvelles découvertes sur les bactéries résistantes aux antibiotiques sont nécessaires de toute urgence. « Le monde est confronté à une crise de résistance aux antibiotiques… S'il faut remonter le temps pour trouver des solutions pour l'avenir, je suis tout à fait pour », a déclaré Michael Mahan, professeur de biologie moléculaire, cellulaire et du développement à l'Université de Californie à Berkeley.
Suggestions de « Jurassic Park »
La plupart des antibiotiques proviennent de bactéries et de champignons, découverts par l'analyse de micro-organismes vivant dans le sol. Mais ces dernières décennies, leur surconsommation a conduit les agents pathogènes à développer une résistance à ces antibiotiques.
Au cours de la dernière décennie, De la Fuente a utilisé des méthodes informatiques pour évaluer le potentiel de divers peptides comme substituts aux antibiotiques. Un jour, au laboratoire, le film à succès « Jurassic Park » est apparu, donnant à l'équipe l'idée d'étudier des molécules disparues. « Pourquoi ne pas ressusciter des molécules du passé ? » a-t-il demandé.
Pour trouver des peptides jusqu'alors inconnus, l'équipe a entraîné un algorithme d'IA à reconnaître des sites fragmentés dans des protéines humaines susceptibles d'avoir une activité antibactérienne. Les scientifiques ont ensuite appliqué cet algorithme à des séquences protéiques accessibles au public provenant d'Homo sapiens, d'Homo neanderthalensis et de Dénisoviens, une autre espèce humaine archaïque étroitement apparentée aux Néandertaliens.
L’équipe a ensuite utilisé les propriétés des peptides antibactériens précédents pour prédire quels peptides anciens étaient les plus susceptibles de tuer les bactéries.
L'équipe a ensuite synthétisé et testé les 69 peptides les plus puissants pour déterminer leur capacité à tuer les bactéries. Elle a sélectionné les six plus puissants, dont quatre provenant de l'Homme moderne, un de Néandertal et un de Dénisoviens.
L'équipe les a exposés à des souris infectées par la bactérie Acinetobacter baumannii, une cause fréquente d'infections nosocomiales chez l'homme. (Une infection nosocomiale est une infection contractée par un patient à l'hôpital, mais qui n'était pas présente lors de son admission.)
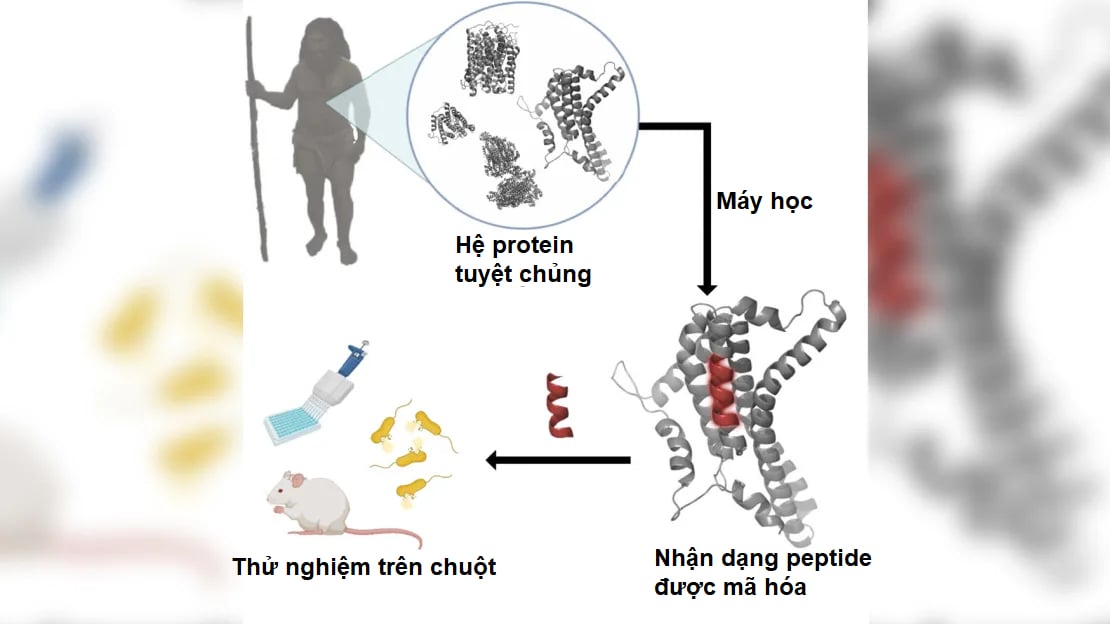
Parmi les six peptides sélectionnés par l'algorithme, celui provenant d'un Néandertalien s'est avéré le plus efficace pour combattre les agents pathogènes chez les souris infectées par des bactéries, a déclaré César de la Fuente, pionnier des biotechnologies de l'Université de Pennsylvanie. (Photo : Université de Pennsylvanie)
« Je pense que l'un des moments les plus passionnants a été celui où nous avons reconstitué chimiquement des molécules en laboratoire, puis les avons ramenées à la vie pour la première fois. C'était extraordinaire d'un point de vue scientifique », a déclaré De la Fuente.
Chez les souris infectées par des abcès cutanés, les peptides ont activement tué les bactéries ; chez les souris atteintes d’infections de la cuisse, les peptides étaient moins efficaces mais empêchaient toujours la croissance bactérienne.
« Le meilleur peptide était ce que nous appelons Neanderthal 1, issu des Néandertaliens, et c’est celui qui fonctionnait le mieux chez la souris », a déclaré De la Fuente.
Des recherches supplémentaires sont nécessaires
Cependant, De la Fuente a souligné qu'aucun de ces peptides n'était « prêt à être utilisé comme antibiotique » et nécessiterait des ajustements importants. Dans une recherche à paraître l'année prochaine, lui et ses collègues ont développé un nouveau modèle d'apprentissage profond pour explorer les séquences protéiques de 208 organismes disparus pour lesquels des informations génétiques détaillées étaient disponibles.
L'équipe a découvert plus de 11 000 peptides antimicrobiens potentiels, jusqu'alors inconnus et présents uniquement chez des créatures disparues, et a synthétisé les peptides les plus prometteurs provenant du mammouth laineux de Sibérie, de la rhytine de Steller (un mammifère marin disparu au XVIIIe siècle à cause de la chasse dans l'Arctique), du paresseux géant et de l'élan géant d'Irlande (Megaloceros giganteus). Il a déclaré que les peptides nouvellement découverts présentaient une « excellente activité anti-infectieuse » chez la souris.
Le Dr Dmitry Ghilarov, chef de groupe au Centre John Innes au Royaume-Uni, a déclaré que le frein à la découverte de nouveaux antibiotiques réside dans leur instabilité et leur difficulté de synthèse. « Nombre de ces antibiotiques peptidiques ne sont ni développés ni exploités par l'industrie en raison de difficultés telles que leur toxicité », a déclaré M. Ghilarov.
Sur les 10 000 composés prometteurs identifiés par les chercheurs, seuls un ou deux antibiotiques ont reçu l'approbation de la Food and Drug Administration américaine, selon un article publié en mai 2021.
Le Dr Monique van Hoek, professeur et directrice associée de recherche à l'École de biologie des systèmes de l'Université George Mason (États-Unis), a déclaré qu'il est très rare qu'un peptide présent dans la nature crée directement un nouveau médicament ou un autre type d'antibiotique.
La découverte d'un nouveau peptide ouvrira la voie aux chercheurs pour utiliser des techniques informatiques afin d'explorer et d'optimiser le potentiel du peptide en tant que nouvel antibiotique, selon Van Hoek.
Van Hoek concentre actuellement ses recherches sur un peptide synthétique dérivé d'un peptide naturel présent chez les alligators américains. Ce peptide est actuellement en phase de tests précliniques.
Bien qu'il puisse paraître étrange de se procurer de nouveaux antibiotiques à partir de crocodiles ou d'humains disparus, la gravité des bactéries résistantes aux antibiotiques rend une telle recherche utile, explique Van Hoek.
Hoai Phuong (selon CNN)
Source




![[Photo] Première séance d'entraînement en préparation du défilé pour célébrer le 80e anniversaire de la Fête nationale, le 2 septembre](https://vphoto.vietnam.vn/thumb/1200x675/vietnam/resource/IMAGE/2025/6/25/ebf0364280904c019e24ade59fb08b18)
![[Photo] Plus de 124 000 candidats à Hanoi ont terminé les procédures pour l'examen de fin d'études secondaires de 2025](https://vphoto.vietnam.vn/thumb/1200x675/vietnam/resource/IMAGE/2025/6/25/fa62985b10464d6a943b58699098ae3f)

![[Photo] Le secrétaire général To Lam travaille avec le Comité permanent des comités provinciaux du Parti de Quang Binh et de Quang Tri](https://vphoto.vietnam.vn/thumb/1200x675/vietnam/resource/IMAGE/2025/6/25/6acdc70e139d44beaef4133fefbe2c7f)




































































































Comment (0)